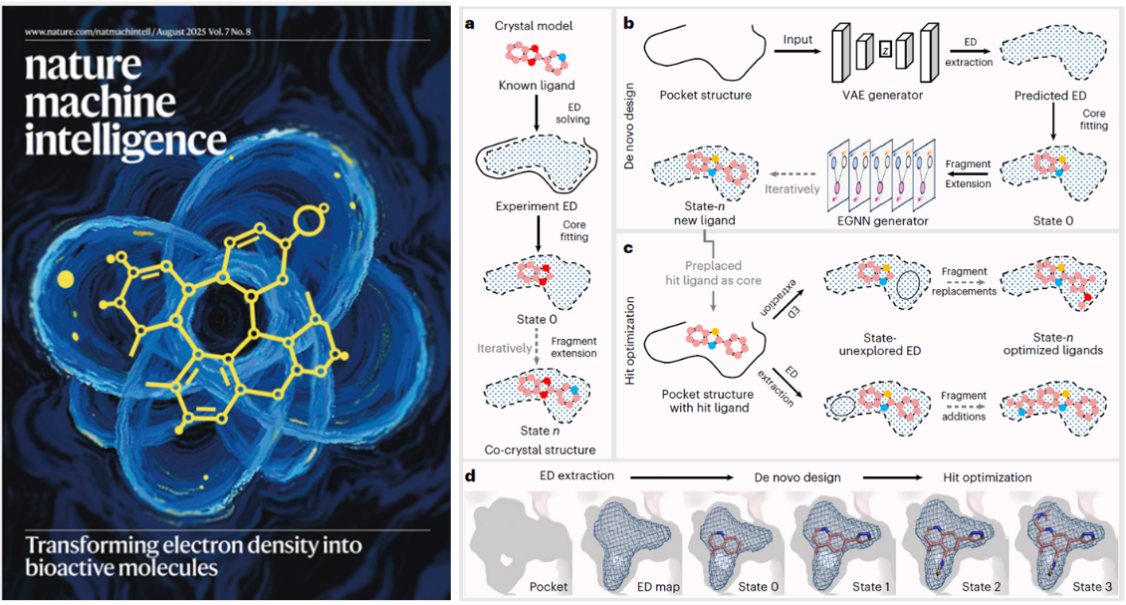
图 人工智能药物设计方法ED2Mol的分子生成架构和工作流程
在国家自然科学基金项目(批准号:81925034, 82441035, 824B2105)等资助下,上海交通大学医学人工智能研究院张健教授课题组,在人工智能生成式药物设计研究方面取得进展。相关成果以“基于电子云密度引导的高效可靠从头分子设计和优化方法ED2Mol(Electron-density-informed effective and reliable de novo molecular design and optimization with ED2Mol)”为题,于2025年8月20日在《自然·机器智能》(Nature Machine Intelligence)杂志在线发表,被该期刊选为封面故事推荐,并获Ana Laura Dias教授在News Highlight专文中的高度评价(https://www.nature.com/articles/s42256-025-01102-x)。论文链接:https://www.nature.com/articles/s42256-025-01095-7。
创新药物研发的核心在于发现具有生物活性的分子(“苗头分子”或“先导分子”)。传统方法主要依赖高通量筛选技术,从现有化合物库中搜寻候选分子。然而,现有化合物库的规模有限(仅约109个分子),结构多样性不足,难以覆盖庞大而复杂的化学空间,使得许多具备潜力的候选分子未被发掘。相比之下,从头设计(de novo)方法直接在估计约1050规模的化学空间中“无中生有”地生成分子,大大提高了发现全新、高效药物分子的可能性。
研究团队开发的电子云密度驱动分子生成模型——ED2Mol,正是这一方向的突破。结构生物学长期以来依赖电子密度图解析分子结构:它不仅宏观展示配体与靶点蛋白的空间匹配,更在微观层面反映原子间的电磁相互作用。ED2Mol首次将电子密度信息融入深度学习生成框架,既能把握结合口袋的整体结构环境,又可精准模拟原子级别的相互作用机制。在多项系统评估中,ED2Mol所生成的分子在结合能力、构象质量与稳定性方面均超越现有方法,同时表现出良好的可合成性、类药性以及丰富的结构多样性。研究团队在成纤维细胞生长因子受体3(FGFR3),细胞分裂控制蛋白42同源物(CDC42),葡萄糖激酶(GCK),视黄酸诱导蛋白(GPRC5A)等靶标体系的药物研发场景中验证了ED2Mol的应用潜力。
该研究开发了从苗头分子生成到先导化合物优化的统一智能框架,生成分子在可靠性与创新性上达到当前最优水平。ED2Mol不仅在常规正构位点表现出色,也能针对难以突破的变构位点生成高效分子,同时覆盖抑制剂与激动剂等多种药物类型,为创新药物研发提供了高效、可靠的人工智能驱动平台。